-
Régulation par des ARN non-codants chez les bactéries
-
De nombreuses découvertes récentes ont montré que l'ARN a une grande diversité de fonction. De plus, nous nous attendons à en découvrir de nombreux autres et beaucoup d'efforts et de recherche sont encore nécessaire pour les découvrir et comprendre leur mécanismes.
-
 Nous utilisons des outils informatiques pour découvrir de nouveaux ARN, les étudier et mieux les comprendre. Nous utilisons aussi des approches biochimiques, génétique et de biologie moléculaire. Parmi nos modèles, les bactéries des genre Burkholderia et Pseudomonas sont privilégiés. Ces bactéries sont largement répandues dans l'environnement, mais peuvent parfois causer de sérieuses infections comme pathogènes opportunistes. Nous sommes également intéressés à découvrir des ARN non-codants dans des communautés bactériennes dans divers environnements, via des études métagénomiques, pour nous aider à comprendre comment ces communautés microbiennes interagissent avec leur environnement.
Nous utilisons des outils informatiques pour découvrir de nouveaux ARN, les étudier et mieux les comprendre. Nous utilisons aussi des approches biochimiques, génétique et de biologie moléculaire. Parmi nos modèles, les bactéries des genre Burkholderia et Pseudomonas sont privilégiés. Ces bactéries sont largement répandues dans l'environnement, mais peuvent parfois causer de sérieuses infections comme pathogènes opportunistes. Nous sommes également intéressés à découvrir des ARN non-codants dans des communautés bactériennes dans divers environnements, via des études métagénomiques, pour nous aider à comprendre comment ces communautés microbiennes interagissent avec leur environnement.
- Quelques exemples des ARN noncodants qui nous intéressent inclus:
- MAEB (Metabolism-Associated with Energy in Burkholderiales), de nouvelles variations structurelles de riboswitch pour la S-adénosylméthionine (SAM) ainsi que de potentiels nouveaux riboswitchs qui restent à caractériser.
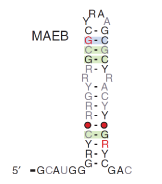
- Nos recherches ne se limitent toutefois pas à quelques espèces. Nous nous intéressons à toutes les espèces bactériennes et aussi d'archaea que nous comparons avec des outils bioinformatiques pour trouver des ARN conservés. Ainsi, nous nous intéressons aussi à des ARN trouvés chez Mycobactérium tuberculosis que nous étudions via une collaboration.
-
Génie métabolique et biologie synthétique
- En parallèle, nous sommes intéressés à utiliser nos connaissances pour faire du génie génétique orienté vers plusieurs types d'applications. Notamment, nous utilisons des petits ARN pour contrôler l'expression des gènes de façons à pousser les bactéries à produire des molécules d'intérêt. À cet effet, nous collaborons avec des compagnies qui utilisent les bactéries comme des "usines cellulaires" pour la production de composés organiques à grande échelle.
-
Aptamères et biosenseurs
- Les aptamères sont des acides nucléiques capables de lié et reconnaître spécifiquement une molécule donnée. Il en existe des versions naturelles (les riboswitchs), mais ils peuvent aussi être sélectionné via une approche d'évolution accélérée in-vitro nommée SELEX. Ainsi, les aptamères peuvent se prêter à une multitude d'applications où la reconnaissance d'une molécule est souhaitée. Notamment, en combinant des aptamères-ARN avec un système d'expression pour en faire des riboswitchs artificiels, ils peuvent être utilisés pour faire des bactéries biosenseurs.
- Comme les aptamères synthétiques peuvent être sélectionnés contre n'importe quel type de cibles (métabolites, contaminants chimiques, protéines ou même des cellules) et ensuite utilisés soit comme "biosenseurs" soit comme "modules de ciblage" pour des particules multitâches, nous avons aussi plusieurs projets en collaboration où des aptamères sont conjugués tantôt à des nanoparticules, tantôt à des fibres optiques, ou encore à d'autres types de molécules comme des peptides dans des perspectives thérapeutiques.
 Nous utilisons des outils informatiques pour découvrir de nouveaux ARN, les étudier et mieux les comprendre. Nous utilisons aussi des approches biochimiques, génétique et de biologie moléculaire. Parmi nos modèles, les bactéries des genre Burkholderia et Pseudomonas sont privilégiés. Ces bactéries sont largement répandues dans l'environnement, mais peuvent parfois causer de sérieuses infections comme pathogènes opportunistes. Nous sommes également intéressés à découvrir des ARN non-codants dans des communautés bactériennes dans divers environnements, via des études métagénomiques, pour nous aider à comprendre comment ces communautés microbiennes interagissent avec leur environnement.
Nous utilisons des outils informatiques pour découvrir de nouveaux ARN, les étudier et mieux les comprendre. Nous utilisons aussi des approches biochimiques, génétique et de biologie moléculaire. Parmi nos modèles, les bactéries des genre Burkholderia et Pseudomonas sont privilégiés. Ces bactéries sont largement répandues dans l'environnement, mais peuvent parfois causer de sérieuses infections comme pathogènes opportunistes. Nous sommes également intéressés à découvrir des ARN non-codants dans des communautés bactériennes dans divers environnements, via des études métagénomiques, pour nous aider à comprendre comment ces communautés microbiennes interagissent avec leur environnement.
